Que lo simple no se vuelva complejo: Para entender las variaciones en SARS-CoV2
Ante la incertidumbre del regreso al semáforo rojo (el caso de México), así como de una vacuna cada vez más próxima, la información relativa a COVID-19 se vuelve de gran importancia. Sin embargo, muchas veces los medios de comunicación logran preocupar a la población general con algunos comunicados insidiosos con poco o nulo sustento científico.
En los últimos días han rondado noticias sobre una “nueva” cepa del SARS-CoV2 en el Reino Unido, lo que ha causado confusión en varias personas debido al enfoque exagerado en las noticias. Por lo cual, en este breve escrito trataré de despejar y aclarar algunas cuestiones del tema.
¿Qué es una cepa?
Se le conoce como cepa al conjunto de variaciones en el material genético de un organismo, que le otorga algunas características particulares y distintas al grupo original. Sin embargo, en el sentido estricto de la definición, la “nueva” cepa de SARS-CoV2 todavía no es considerada una cepa distinta, debido a que los cambios en su genoma, todavía no demuestran causalmente una mayor letalidad, contagio u otra característica distinta al espécimen identificado en el mercado de Wuhan.
Sí no es cepa, entonces ¿qué es?
Tal como en todos los seres biológicos, también existe variación en los virus. Las variaciones o mutaciones del virus son diversas, y en muchos casos son al azar en su material genético. Aunque podrían ser direccionadas según ciertos fenómenos evolutivos con un lapso de tiempo mayor, sin embargo, esto no es tan relevante en estos momentos.
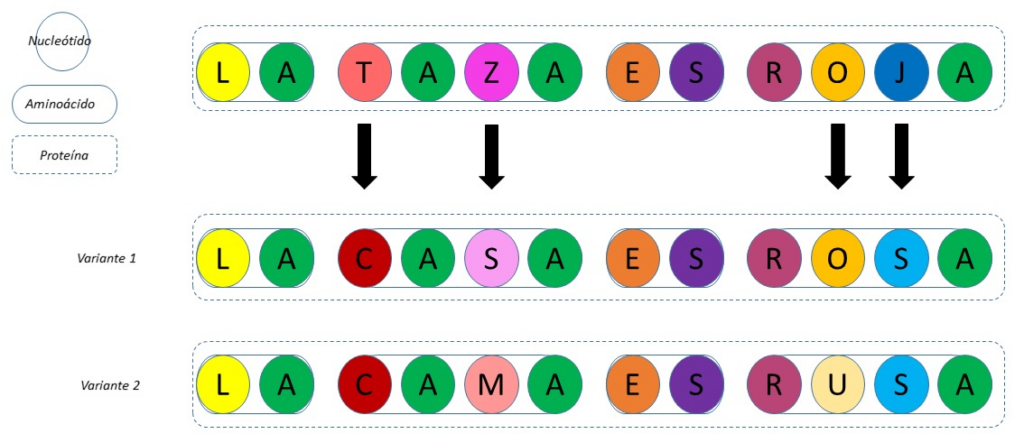
Para comprender el fenómeno de variación del virus de forma más sencilla, podemos citar el siguiente ejemplo: La frase “La taza es roja” sería equivalente a una proteína, formada por aminoácidos (palabras), y estos a su vez, formados por nucleótidos (letras). En un principio, la frase sería conocida y entendida por todos, sin embargo, el mensaje podría cambiar -por cuestiones del azar- a “La taza es rosa”, es decir, en una de sus letras (nucleótidos), y en consecuencia la palabra (aminoácido) en donde se encontraba la misma, para finalmente cambiar el sentido de roja a rosa. De esta forma podían suceder cambios en cualquier letra, lo que modificaría cualquiera de las palabras que componen la frase (proteína), y finalmente el sentido de la frase. Biológicamente, este proceso sucede todo el tiempo originando variaciones entre una misma especie, sin embargo, los cambios significativos llegan a suceder en tiempos sumamente amplios al tiempo humano habitual.
Estas mutaciones suceden al azar y se calculan con distinta rapidez (tasa) de cambio. En general, los virus de ARN como el SARS-CoV2, muestran cambios más rápidos que los de ADN, por lo que se explicaría su relativamente rápida transformación. Se ha dirigido la atención a ciertas mutaciones identificadas en regiones particulares del mensaje inicial que alterarían algunos aminoácidos y con ello quizá algunas proteínas cruciales en el virus. Tal como la proteína S con las variaciones D614G, N501Y o P681H (nomenclatura para los cambios de aminoácido), en la que se han observado sus efectos en algunos experimentos, sucediendo independientemente, y aumentando la preocupación que lleguen a suceder en una misma variante. Por este motivo, se ha pedido el confinamiento y limitación de viajes internacionales para evitar la posible “mezcla” de distintas variantes, hecho que hasta ahora no se ha documentado, pero es probable que pueda ocurrir. Siguiendo el ejemplo del inicio, de trascender esta mezcla de variantes podría degenerarse el mensaje hasta resultar en “La casa es rosa” o “La casa es rusa”, es por ello que debemos tomar con responsabilidad las salidas esenciales solamente.
¿Qué significa que existan otras variantes del SARS-CoV2?
La identificación de distintas variantes alrededor del mundo han sido reportadas desde el inicio de la misma pandemia, debido a la excelente iniciativa de liberar los datos de las secuencias genéticas en repositorios libres al alcance de todos los grupos científicos. 1–3 Estas variantes puntuales se han ido agrupando en subgrupos o clados del grupo original, de los cuales se han reportado en varias regiones geográficas del mundo. Hasta ahora se han reportado algunas diferencias en la tasa de mortalidad de cada uno de estos clados, aunque es de baja probabilidad que en un periodo corto de tiempo estas mutaciones tengan una repercusión significativa en la estructura genética del virus, cambiando estructuralmente una proteína y haciéndola potencialmente más o menos infectiva según sea el caso.
La variante identificada en el Reino Unido, denominada B.1.1.7, se ha reportado que posee 8 mutaciones de interés en la sección que codifica a la proteína S, lo que ha activado las alarmas en esos países -al grado de cerrar vías de acceso- a pesar de que todavía no se tiene una conclusión certera de si es más infectiva o mortal. 4,5 El cierre parecería ser una medida cautelosa y bastante racional, debido a que SARS-CoV2 ha demostrado romper distintos paradigmas científicos en todo el mundo.
Respecto a las vacunas en las distintas fases de investigación clínica, así como tipos de vector o vehículo utilizado, se ha reportado que independiente de la variación del SARS-CoV2, es muy probable de que sea efectiva en todas las variantes hasta ahora reportadas, por lo que podemos estar tranquilos ante este fenómeno. 6
La respuesta para frenar esta pandemia es simple con todas las medidas que se han implementado y en conjunto con la vacuna, sin embargo, ha sido bastante compleja de llevar a cabo por cuestiones más allá de la biología. Retomando el ejemplo inicial, está en nuestras acciones y comportamientos en dejar que el mensaje permanezca tan simple como la taza es roja, y no se torne compleja y rebuscada con sentidos borgesianos o cortazarianos.
Referencias
1. Lu R, Zhao X, Li J, et al. Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding. Lancet. 2020;395(10224):565-574. doi:10.1016/S0140-6736(20)30251-8.
2. Koyama T, Platt D, Parida L. Variant analysis of SARS-cov-2 genomes. Bull World Health Organ. 2020;98(7):495-504. doi:10.2471/BLT.20.253591.
3. Khailany RA, Safdar M, Ozaslan M. Genomic characterization of a novel SARS-CoV-2. Gene Reports. 2020;19:100682. doi:10.1016/j.genrep.2020.100682.
4. Rambaut A, Loman N, Pybus O, et al. Preliminary genomic characterisation of an emergent SARS-CoV-2 lineage in the UK defined by a novel set of spike mutations – SARS-CoV-2 coronavirus / nCoV-2019 Genomic Epidemiology – Virological. https://virological.org/t/preliminary-genomic-characterisation-of-an-emergent-sars-cov-2-lineage-in-the-uk-defined-by-a-novel-set-of-spike-mutations/563. Published 2020.
5. European Centre for Disease Prevention and Control. Rapid Increase of a SARS-CoV-2 Variant with Multiple Spike Protein Mutations Observed in the Rapid Increase of a SARS-CoV-2 Variant with Multiple Spike Protein Mutations Observed in the United Kingdom.; 2020. http://covid19-country-overviews.ecdc.europa.eu/#34_United_Kingdom.
6. Dearlove B, Lewitus E, Bai H, et al. A SARS-CoV-2 vaccine candidate would likely match all currently circulating variants. Proc Natl Acad Sci U S A. 2020;117(38):23652-23662. doi:10.1073/pnas.2008281117.




Comentarios recientes